Modul Praktikum Sistematika Mikrobia
Departemen Biologi, Fakultas Sains dan Analitika Data, Institut Teknologi Sepuluh Nopemberenny@bio.its.ac.id Departemen Biologi, Fakultas Sains dan Analitika Data, Institut Teknologi Sepuluh Nopember
danilyanedo7@gmail.com Departemen Biologi, Fakultas Sains dan Analitika Data, Institut Teknologi Sepuluh Nopember
sabilafatimah@gmail.com
2022-04-08
1 TAKSONOMI NUMERIK FENETIK
1.1 Klasifikasi Numerik-Fenetik Berdasarkan Data Fenotipik
1.1.1 Pengantar
Taksonomi numerik yang juga dikenal dengan sebutan taksonomi Adansonian (berdasarkan nama ahli sistematika Michael Adanson) didefinisikan sebagai pengelompokan unit taksonomis ke dalam sejumlah taksa dengan metode numerik berdasarkan karakteristik yang dimiliki. Taksonomi numerik didasarkan atas lima prinsip utama, yakni:
- Taksonomi yang ideal adalah taksonomi yang mengandung informasi terbesar yaitu yang didasarkan atas sebanyak-banyaknya karakter.
- Masing-masing karakter diberi nilai yang setara (a priori) dalam mengkonstruksi takson yang bersifat alami.
- Tingkat kedekatan antara dua strain/OTU (operational taxonomical unit) merupakan fungsi similaritas yang dimiliki bersama.
- Taksa yang berbeda dibentuk atas sifat yang dimiliki.
- Similaritas tidak bersifat filogenetis melainkan bersifat fenetis.
Tujuan utama taksonomi numerik adalah untuk menghasilkan suatu klasifikasi yang bersifat teliti, reprodusibel, serta padat informasi. Aplikasi taksonomi numerik dalam konstruksi klasifikasi biologis memungkinkan terwujudnya sirkumskripsi takson berdasarkan prinsip yang mantap dan bukan sekedar klasifikasi yang bersifat subjektif belaka. Teknik klasifikasi meliputi empat tahapan, yaitu:
- Strain mikrobia (n) yang akan diklasifikasikan dikoleksi lalu ditentukan karakter fenotipiknya dalam jumlah besar (t) yang mencakup sifat yang tertera pada Tabel 1.1. Data yang diperoleh disusun dalam suatu matriks n x t.
- Strain mikrobia diklasifikasikan berdasarkan nilai similaritas atau disimilaritas yang dihitung dari data n x t.
- Strain yang mirip akan dimasukkan ke dalam satu kelompok dengan menggunakan algoritma pengklasteran (clustering algorithm).
- Kelompok yang dibentuk secara numerik lalu dipelajari dan karakter yang bersifat membedakan (separating character) dipilih diantara data dalam matriks untuk selanjutnya digunakan dalam dentifikasi.
1.1.2 Cara Kerja
Tahapan kerja dalam klasifikasi numerik-fenetik terdiri dari (1) pemilihan strain uji, (2) pemilihan jenis pengujian, (3) pencatatan hasil pengujian, (4) data coding, (5) analisis komputer, dan (6) interpretasi hasil (Priest & Austin, 1993). Namun demikian, dalam praktikum ini kita akan menggunakan data hasil pengujian dari artikel penelitian yang terdapat pada jurnal ilmiah dalam bentuk tabel n x t. Namun, format yang digunakan adalah tidy data, yaitu setiap kolom merepresentasikan karakter (t) sementara setiap baris merepresentasikan sampel mikroorganisme(n). Dengan demikian, kita telah melewati tahapan kerja 1–3 dan langsung menuju tahapan data coding dan seterusnya.
Tabel 1.1. Kelompok karakter yang digunakan dalam taksonomi numerik bakteri (Priest & Austin, 1993).
No | Kelompok Karakter | Jenis Karakter |
1 | Morfologi kolonial | Bentuk dan ukuran koloni, pigmentasi larut/tidak larut/ |
fluoresens. | ||
2 | Morfologi selular | Sifat pengecatan, bentuk & ukuran sel, motilitas, |
ada/tidak nya flagela, ada/tidaknya spora. | ||
3 | Sifat pertumbuhan | Bentuk-bentuk pertumbuhan pada medium cair, kekeruhan, aerobsis/anaerobsis, kebutuhan vitamin, |
kemampuan tumbuh pada medium salinitas tinggi. | ||
4 | Sifat biokimia | Kemampuan fermentasi/oksidasi karbohidrat, kehadiran enzim katalase & oksidase, penghasilan asam, indol, & gas H2S |
5 | Resistensi terhadap antibiotik | Kemampuan untuk tumbuh pada medium yang mengandung antibiotik |
6 | Kemampuan penggunaan senyawa kimia sebagai satu- satunya sumber C | Kemampuan tumbuh pada medium minimal dengan glukosa/alanin/sitrat sebagai satu-satunya sumber karbon |
7 | Sifat serologis | Ada/tidaknya reaksi aglutinasi terhadap antiserum spesifik |
8 | Sifat kemotaksonomis | Kehadiran komponen sub-selular tertentu, seperti peptidoglikan, menakuinon, asam mikolat. |
9 | Sifat genetik | Kandungan G+C pada DNA |
10 | Phage typing | Ada/tidaknya pola phage typing tertentu |
1.1.2.1 Penelusuran Sumber Data
Dalam praktikum ini, data untuk klasifikasi numerik-fenetik didapatkan melalui hasil penelusuran artikel ilmiah. Artikel ilmiah tersebut dapat dicari dari internet melalui Google Search dengan mengetikan kata-kata kunci terkait klasifikasi numerik fenetik seperti contohnya “numerical taxonomy of…” atau “numerical classification of…” Salah satu contoh artikel dapat dilihat pada West et al. (1986) yang mempelajari klasifikasi spesies anggota genus Vibrio yang diisolasi dari perairan. Data yang terdapat pada artikel tersebut selanjutnya akan dijadikan contoh pada panduan praktikum ini. Setelah mendapatkan artikel jurnal, telusuri tabel n x t yang terdapat di dalam artikel tersebut. Walaupun sering tidak disebut sebagai tabel n x t, namun tabel n x t dapat terlihat dari cirinya yang mengandung informasi mengenai strain-strain mikrobia (n) serta jenis karakter yang diujikan (t). Karakter yang terkandung dalam tabel n x t pada umumnya akan dikelompokan seperti pada Tabel 1.1
1.1.2.2 Konstruksi Tabel
Berdasarkan tabel n x t yang diperoleh dari artikel jurnal penelitian, selanjutnya kita membuat tabel n x t sendiri. Tabel n x t dapat dibuat dengan program Microsoft Excel (MS Excel). Pada MS Excel, strain (n) dimasukan sebagai baris dan karakter pengujian (t) dimasukan sebagai kolom (Gambar 1.1.). Pengkodean pada tabel n x t menggunakan sistem biner, yakni notasi “1” dan “0”. Notasi “1” diberikan apabila ada kehadiran suatu sifat dan notasi “0” diberikan untuk ketidakhadiran suatu sifat. Jika dilihat pada Gambar 1 tersebut, kemampuan V. fluvialis untuk dapat tumbuh pada medium dengan kandungan NaCl 6% menjadikan spesies tersebut mendapatkan notasi “1” pada karakter “Pertumbuhan 6% NaCl”. Sebaliknya, V. fluvialis tidak dapat tumbuh pada medium dengan kandungan NaCl 0% sehingga membuatnya mendapatkan notasi “0” untuk sifat tersebut. Dalam laporan tertulis atau publikasi ilmiah, penggunaan notasi “1” dan “0” pada n x t sebagai indikasi kehadiran dan ketidakhadiran suatu sifat pada suatu strain terkesan kurang lazim. Pada umumnya, notasi “+” dan “–“ digunakan sebagai pengganti “1” dan “0”. Namun demikian, notasi “1” dan “0” pada n x t dimaksudkan untuk kepentingan analisis menggunakan program komputer, karena program tersebut tidak dapat mengenali notasi “+” dan “–“.
## New names:
## * `` -> ...1...1 | UF1 | UF4 | UF5 | UF6 | UF8 |
Circular | 1 | 0 | 1 | 0 | 0 |
Rhizoid | 0 | 1 | 0 | 0 | 0 |
Irregular | 0 | 0 | 0 | 1 | 1 |
Flat | 0 | 1 | 0 | 0 | 0 |
Convex | 0 | 0 | 1 | 0 | 0 |
Raised | 1 | 0 | 0 | 1 | 1 |
Entire | 1 | 0 | 1 | 0 | 0 |
Undulate | 0 | 0 | 0 | 0 | 0 |
Filamentous | 0 | 1 | 0 | 0 | 0 |
Erose | 0 | 0 | 0 | 1 | 1 |
Opaque | 1 | 0 | 1 | 0 | 0 |
Translucent | 0 | 1 | 0 | 1 | 1 |
Shiny | 1 | 0 | 1 | 0 | 0 |
Dull | 0 | 1 | 0 | 1 | 1 |
Yellow | 0 | 0 | 0 | 0 | 0 |
Cream | 0 | 0 | 1 | 0 | 0 |
White | 1 | 1 | 0 | 1 | 1 |
Smooth | 1 | 0 | 1 | 0 | 0 |
Rough | 0 | 1 | 0 | 1 | 1 |
Sel batang | 1 | 1 | 1 | 1 | 1 |
Gram | 1 | 1 | 1 | 1 | 1 |
Endospora | 1 | 1 | 1 | 1 | 1 |
Aerob obligat | 1 | 0 | 1 | 1 | 1 |
Fakultatif anaerob | 0 | 1 | 0 | 0 | 0 |
Uji Motilitas | 1 | 1 | 1 | 1 | 1 |
Uji Katalase | 1 | 1 | 1 | 1 | 1 |
Uji Hidrolisis Amilum | 0 | 1 | 1 | 1 | 0 |
Uji Hidrolisis Gelatin | 0 | 0 | 0 | 0 | 0 |
Uji Hidrolisis Kasein | 0 | 0 | 1 | 1 | 0 |
Uji Sitrat | 0 | 0 | 1 | 0 | 0 |
Uji Toleransi Salinitas NaCl 3% | 1 | 1 | 1 | 1 | 1 |
Uji Toleransi Salinitas NaCl 6,5% | 0 | 0 | 1 | 1 | 0 |
Uji Toleransi Salinitas NaCl 10% | 0 | 0 | 1 | 0 | 0 |
Uji VP | 0 | 1 | 1 | 0 | 1 |
Uji MR-VP | 1 | 1 | 1 | 1 | 1 |
Uji Fermentasi Glukosa menghasilkan asam | 1 | 1 | 1 | 1 | 1 |
Uji Fermentasi Glukosa menghasilkan gas | 0 | 0 | 0 | 0 | 0 |
Uji Fermentasi Arabinosa menghasilkan asam | 0 | 0 | 0 | 0 | 0 |
Uji Fermentasi Arabinosa menghasilkan gas | 0 | 0 | 0 | 0 | 0 |
Diameter sel ≥ 1µ | 1 | 1 | 1 | 1 | 1 |
Uji Resisten Fe 30 ppm | 1 | 1 | 1 | 1 | 1 |
Uji Resisten Fe 90 ppm | 1 | 1 | 1 | 1 | 1 |
Uji Resisten Fe 120 ppm | 1 | 1 | 1 | 1 | 1 |
Uji Resisten Fe 350 ppm | 0 | 0 | 0 | 0 | 0 |
Resisten suhu 80◦ | 1 | 1 | 1 | 1 | 1 |
Perlu diketahui bahwa pemilihan spesies dalam konstruksi tabel n x t sebaiknya mencakup type strain untuk spesies tersebut. Dengan kata lain, sangat disarankan apabila data yang digunakan juga memiliki hasil pengujian untuk type strain. Suatu type strain merupakan strain yang mewakili suatu spesies atau dengan kata lain type strain merupakan pemilik nama spesies. Kehadiran suatu type strain dalam proses konstruksi dendrogram berfungsi sebagai acuan bagi strain-strain lainnya yang memiliki kesamaan nama spesies dengan type strain, sehingga dapat lebih meyakinkan posisi strain-strain tersebut dalam suatu dendrogram
1.1.2.3 Konstruksi Dendogram Menggunakan Bahasa Pemrograman R
library(tidyverse)## -- Attaching packages --------------------------------------- tidyverse 1.3.1 --## v ggplot2 3.3.5 v purrr 0.3.4
## v tibble 3.1.6 v dplyr 1.0.8
## v tidyr 1.2.0 v stringr 1.4.0
## v readr 2.1.2 v forcats 0.5.1## -- Conflicts ------------------------------------------ tidyverse_conflicts() --
## x purrr::compose() masks flextable::compose()
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()library(factoextra)## Welcome! Want to learn more? See two factoextra-related books at https://goo.gl/ve3WBa# Input data secara manual menggunakan fungso data.frame
mie_instan <- data.frame(nama = c("Indomie rendang", "Indomie soto", "Indomie kari", "Sedap goreng", "Sedap soto", "sedap kari"),
kuah = c(0, 1, 1, 0, 1, 1),
garam = c(1.5, 1.4, 1.5, 1.8, 2, 2.1),
fiber = c(10, 12, 12, 15, 15, 15),
kalori = c(1015, 1016, 1017, 1700, 1750, 1690))
# Input data dari sistem peyimpanan lokal
contoh_data <- readxl::read_excel("C:/Users/Lenovo/Documents/praktikumsismikbits/data/contohdatatidy.xlsx")
View(contoh_data)## Warning in do.call(data.frame, c(x, alis)): unable to translate 'Resisten suhu
## 80<U+25E6>' to native encodingdatamikro <- contoh_data |> column_to_rownames(var = "0")
dist_mikro <- dist(datamikro, method = 'euclidean')
clust_mikro <- hclust(dist_mikro, method = "average")
plot(clust_mikro)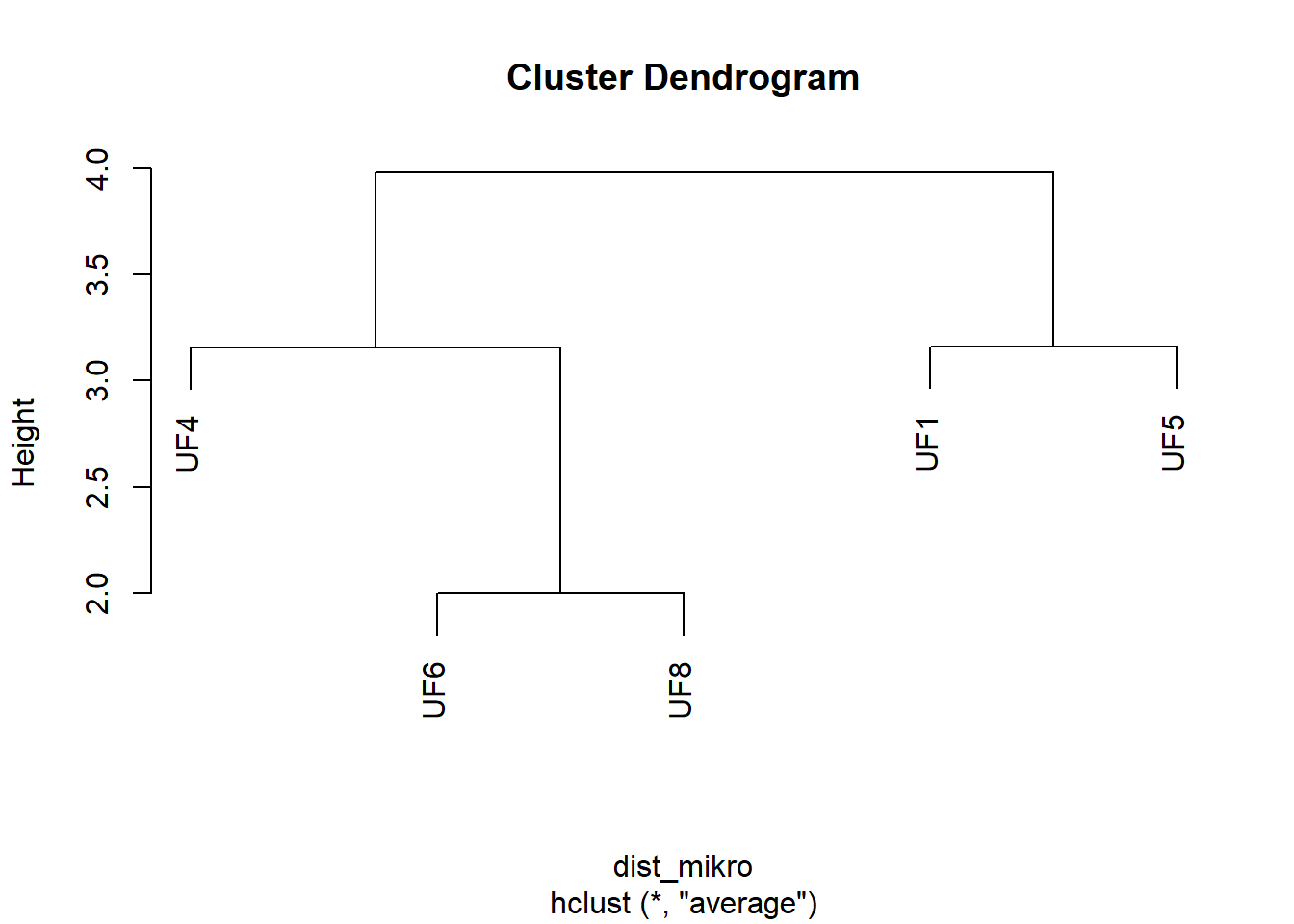
1.2 Klasifikasi Numerik-Fenetik Berdasarkan Profil Sidik Jari Molekular
1.2.1 Pengantar
Selain data fenotipik, data berupa profil sidik jari molekular (molecular fingerprints) juga dapat digunakan dalam studi taksonomi mikrobia. Profil yang digunakan dapat berasal dari DNA, RNA, atau protein. Studi klasifikasi menggunakan profil sidik jari molekular termasuk ke dalam studi kemosistematik bersama dengan analisis komponen selular lainnya (Priest & Austin, 1993). Teknik yang paling umum digunakan dalam studi klasifikasi berdasarkan profil sidik jari molekular adalah restriction fragment length polymorphisms (RFLP). Prinsip dasar dari penggunaan RFLP adalah adanya kesamaan situs restriksi antara satu mikroorganisme dengan mikroorganisme lainnya yang dapat dikenali oleh enzim restriksi endonuklease tertentu. Hal ini berarti bahwa distribusi situs- situs restriksi tersebut dapat memberi gambaran mengenai kemiripan antara satu mikroorganisme dengan lainnya secara molekular.
1.2.2 Cara Kerja
Seperti halnya pada praktikum acara 1, data yang dipakai dalam praktikum acara 2 ini diperoleh dari artikel penelitian yang terdapat pada jurnal ilmiah dalam bentuk elektroforegram (foto elektroforesis).
Penelusuran Sumber Data Data yang digunakan dalam praktikum ini berupa elektroforegram sidik jari (fingerprint) molekular. Data fingerprint tersebut dapat berupa hasil ribotyping, RFLP, RAPD, dan lainnya. Elektroforegram dari suatu artikel jurnal dapat di-crop menggunakan program PDF reader seperti Adobe Reader melalui menu Tools → Select & Zoom → Snapshot Tool. Buat file gambar (image file) format JPEG atau PNG dari elektroforegram tersebut dengan memblok elektroforegram yang diinginkan dan copy (ctrl+C) ke program Paint.
Pembuatan Diagrammatic Representative Diagrammatic representative merupakan sebuah gambaran skematis yang mewakili elektroforegram yang dianalisis. Penggunaan diagrammatic representative ini barfungsi untuk mempertegas kehadiran atau ketidakhadiran suatu band dalam elektroforegram. Kejelasan mengenai hadir atau tidaknya sebuah band merupakan hal yang penting karena band tersebut diasumsikan sebagai karakter dalam data coding.
Pembuatan diagrammatic representative dapat dilakukan dengan program photo editor seperti Corel Draw, Adobe Photoshop, atau Paint Shop Pro. Secara umum, tahapan pembuatannya meliputi input gambar, penambahan layer transparan, dan menggambarkan suatu garis pada layer transparan tersebut yang berpandu pada band elektroforegram dibawahnya. Dalam praktikum ini program Paint Shop Pro (PSP) akan digunakan untuk pembuatan diagrammatic representative ini. Pertama, buka program PSP dan masukan file gambar elektroforegram yang telah dibuat sebelumnya melalui menu File → Open. Setelah gambar elektroforegram muncul, akan terdapat suatu kotak bertuliskan Layer pada sisi sebelah kanan. Apabila kotak ini tidak muncul, kita dapat memunculkannya melalui ikon Toggle Layer Palette pada sisi atas, ikon kedua dari kanan. Pada layer palette tersebut, gambar yang kita masukan sebelumnya dikategorikan sebagai Background. Kita dapat menambahkan layer baru dengan menekan ikon Add New Layer yang terletak pada pojok kiri bawah layer palette. Sebuah layer baru yang muncul akan diberi nama Layer 1 secara otomatis dan terletak di atas layer Background. Klik ikon Layer 1 untuk mengaktifkannya dan Pada Layer 1 inilah kita akan membuat diagrammatic representative dari elektroforegram.
Setelah memilih Layer 1, aktifkan control palette dengan menekan ikon Toggle Control Palette yang terletak 3 ikon di sebelah kiri Toggle Layer Palette. Menu control palette berfungsi untuk mengatur jenis, ketebalan, bentuk, dan ukuran brush tip yang akan digunakan untuk mewarnai Layer 1 berdasdarkan cetakan yang ada pada elektroforegram. Sebelum mengatur apapun yang ada pada control palette, aktifkan Paint Brushes yang terletak pada tool palette (menu di pojok kiri). Pemilihan warna dapat dilakukan pada color palette (menu di pojok kanan). Kedua menu tersebut masing-masing dapat diaktifkan melalui Toggle Tool Palette dan Toggle Color Palette. Menu Control Palette terdiri atas dua submenu, yakni Tool Controls dan Brush Tip. Untuk sementara, abaikan dahulu submenu Tool Controls dan masuk ke submenu Brush Tip. Pada submenu Brush Tip terdapat pilihan Shape, Size, Opacity, Hardness, Density, dan Step yang masing-masing berfungsi untuk mengatur jenis kuas, ukuran kuas, ketebalan warna, ketegasan bentuk kuas, kepadatan warna, dan gradasi kuas. Hasil pengaturan enam parameter ini langsung ditampilkan pada layar di kiri atas submenu Brush Tip. Sebagai contoh, pengaturan Brush Tip yang digunakan meliputi Shape: Horizontal, Opacity: 25, Hardness: 50, Density: 100, dan Step: 1. Size yang digunakan bergantung pada ukuran image file yang digunakan. Sebelum mulai membuat diagrammatic representative, sebaiknya kita mengujikan hasil pengaturan pada submenu Brush Tip untuk melihat kecocokannya langsung pada Layer 1.
Hasil pengujian kemudian dapat dihapus dengan mengaktifkan ikon Eraser pada tool palette. Bentuk dan ukuran Eraser juga diatur pada menu Control Palette yang sama dengan Brush Tip. Hasil penggambaran dapat dilihat pada Gambar 2.1. Setelah penggambaran selesai dilakukan, simpan file tersebut (PSP akan secara otomatis menyimpannya dalam format .psp). File .psp ini juga dapat dibuka dengan program image viewer seperti ACDSee. Diagrammatic representative yang ditampilkan adalah hasil penggambaran yang dilakukan tanpa menyertakan background foto elektroforegram. ((((NOTE GAMBAR))))
Dengan demikian, kita dapat menghilangkan foto elektroforegram dengan meng-klik ikon Background pada Layer Palette dan kemudian Delete. Hasilnya hanya akan tersisa pita-pita berwarna hasil penggambaran sebelumnya (Gambar 2.2). Simpan kembali file ini dalam format .jpg (gunakan menu Save As) dengan nama lain agar tidak mengganti file sebelumnya.
- Data Coding dan Analisis Diagrammatic representative yang dihasilkan merupakan sumber untuk data coding. Dalam hal ini, setiap pita yang tergambarkan pada diagrammatic representative merupakan karakter, sehingga total seluruh pita dari seluruh strain mencerminkan jumlah karakter (t) pada tabel n x t. Konversi diagrammatic representative menjadi tabel n x t dapat dilakukan secara intuitif dengan melihat kesejajaran pita antar strain dan menggolongkan pita-pita yang sejajar sebagai satu karakter (Gambar 2.3). Namun demikian, apabila jumlah pita terlalu banyak maka diperlukan garis bantu untuk melihat kesejajaran antar pita. Garis bantu tersebut dapat dibuat pada program Paint. Buka file gambar diagrammatic representative dalam format .jpg dengan program Paint dan klik ikon Line yang terletak pada bagian atas. Setelah itu buat garis dari ujung kiri ke ujung kanan gambar dimulai dari pita teratas. Pita-pita yang bersinggungan dengan garis tersebut dinyatakan sejajar dan menempati karakter yang sama.
Tabel n x t yang telah dibuat selanjutnya dianalisis secara numerik-fenetik sama seperti pada analisis data fenotipik. Analisis dilakukan menggunakan program MVSP 3.1 (Kovach, 2007) dengan indeks similaritas SSM dan SJ serta algoritme pengklasteran UPGMA.
- Interpretasi Hasil Hasil analisis numerik-fenetik data fingerprint molekular ini sekilas terlihat sama dengan data fenotipik, yakni terdiri atas matriks similaritas, analisis klaster, dan dendrogram. Akan tetapi, perlu diperhatikan bahwa kaidah yang diterapkan pada data fenotipik tidak harus diterapkan pada data fingerprint ini. Salah satu syarat seperti digunakannya minimal 50 karakter pada data fenotipik tidak perlu diterapkan pada data fingerprint. Hal ini didasarkan pada fakta bahwa profil fingerprint yang ada dipandang hanya sebagai salah satu karakter saja dan tabel n x t yang dikonstruksi merupakan turunan dari satu karakter tersebut.